仅根据蛋白质序列便可预测相互作用界面,一种基于Transformer的蛋白预测模型
第一时间掌握
蛋白质-蛋白质相互作用(PPI)对于生物过程至关重要,预测这些相互作用的位点对于计算和实验应用都很重要。
杜克大学(DukeUniversity)和康奈尔大学(CornellUniversity)的研究人员提出了一种与结构无关的语言Transformer和肽优先级(Structure-agnosticLanguageTransformerandPeptidePrioritization,SaLT&PepPr)管线,用于仅根据蛋白质序列预测相互作用界面,以用于后续生成肽结合基序。
该模型通过每个位置预测任务对ESM-2蛋白质语言模型(pLM)进行微调,从而使用PDB中的数据识别PPI位点,并优先考虑最有可能参与链间结合的基序。通过仅使用氨基酸序列作为输入,该模型与基于结构同源性的方法具有竞争力,但与同时输入结构和序列特征的深度学习模型相比,性能下降。
受使用共晶设计目标结合「引导」肽结果的启发,研究人员整理了PPI数据库来识别后续肽衍生的分子伴侣。将引导肽融合到E3泛素连接酶结构域,该团队证明了内源性β-连环蛋白、4E-BP2和TRIM8的降解,并强调了他们在癌细胞中表现最佳的降解剂的纳摩尔结合亲和力、低脱靶倾向和功能改变能力。
该团队表明,通过pLM优先考虑自然相互作用中的结合物可以实现可编程的蛋白质靶向和调节。
研究「SaLT&PepPrisaninterface-predictinglanguagemodelfordesigningpeptide-guidedproteindegraders」,于2023年10月24日发布在《CommunicationsBiology》。

将紧凑蛋白结合物与各种E3泛素连接酶结构域融合,能够选择性结合、泛素化和不同目的蛋白的细胞内降解。生成一个模块化系统来设计这些基因编码的构建体,称为泛在体(uAb),将代表一种灵活的靶向蛋白质降解(TPD)方法。
受到RNA引导的CRISPR基因组编辑的可编程性的启发,杜克大学和康奈尔大学的研究人员之前使用从蛋白质-蛋白质相互作用(PPIs)的结合共晶结构的结合界面中鉴定出的线性基序作为后续生成靶标降解uAb的「引导」肽。
然而,这种基于结构的方法依赖于经过实验验证的目标蛋白共晶,这种共晶仅占人类蛋白质组的<25%。金标准PPI数据库包含超过75%的人类蛋白质组的结合序列,因此代表了指导肽生成的丰富信息来源。
因此,研究人员假设利用PPI信息从伴侣蛋白序列中识别蛋白相互作用位点,可能能够对uAb介导的TPD的引导肽进行更广泛的优先排序。
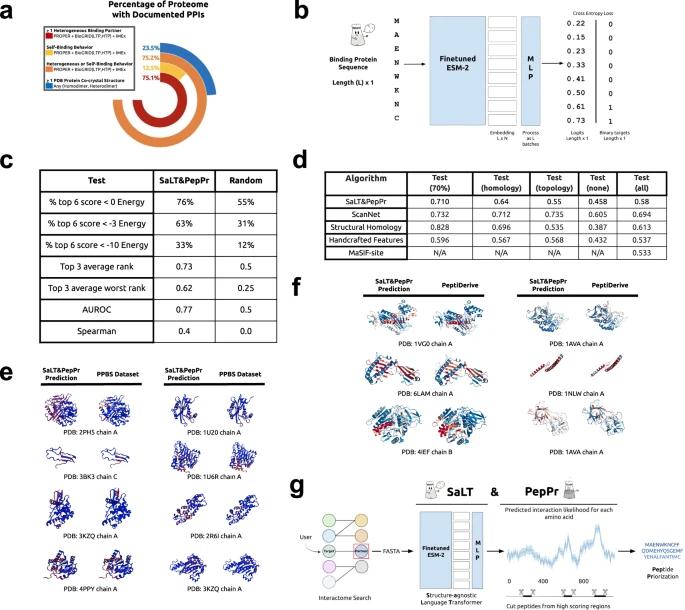
在最新的工作中,该团队应用蛋白质语言模型(pLM)来识别输入蛋白质序列中的结合基序,而不需要三维蛋白质结构。通过准确预测经过验证的相互作用伙伴上的这些蛋白质结合位点,他们优先考虑用于下游uAb生成的引导肽。
为此,该团队基于最先进的ESM-2pLM创建了一个与结构无关的语言Transformer和肽优先级(SaLT&PepPr)模型,首先预测沿输入相互作用伙伴序列的相互作用位点,并通过与PPI数据库集成,实现输入靶蛋白的连续引导肽候选物的分离。
作为第一个概念验证,研究人员利用已知的相互作用信息来生成高亲和力、特异性肽引导的β-连环蛋白降解剂,β-连环蛋白是一种核心转录调节因子,其失调经常导致癌细胞增殖。
然后,研究人员证明SaLT&PepPr可以以数据驱动的方式有效地将引导肽优先考虑到4E-BP2和TRIM8,这些引导肽被集成到uAb架构中,并发现可以诱导靶标降解。具体来说,靶向TRIM8的uAb可诱导尤文肉瘤细胞凋亡,这与之前的遗传学研究一致。
该研究进一步证明了pLM在识别蛋白质-蛋白质结合界面方面的应用,虽然它对结构化目标的效果不如当前最先进的方法,但与结构同源性或基于特征的方法相比具有竞争力。研究人员设想未来的模型将更大的语言模型与进化和结构信息相结合,并直接考虑相互作用的蛋白质,可能会进一步提高计算和实验性能。
总的来说,通过将基于pLM的结合预测与用于蛋白质降解的uAb相结合,该工作激发了利用蛋白质相互作用来设计用于大规模蛋白质组编辑应用的可编程工具。
论文链接:https://www.nature.com/articles/s42003-023-05464-z
